Mapchart绘制基因染色体位置图
最近,MapGene2Chrom web v2(http://mg2c.iask.in/mg2c_v2.0/)偶有“歇菜”,让我们想简单的画画基因的染色体位置图都不方便。在这里,小编给 大家介绍一个比较早的软件Mapchart,来帮助我们解决这一烦恼。
Mapchart是Windows系统下,进行基因在染色体上定位图片绘制的工具,主要是针对遗传连锁图和QTL。区别于遗传图谱中遗传距离,我们这里直接可以得到基因在染色体上的物理位置信息,也可以利用Mapchart画图。
软件Mapchart
下载地址:
https://www.wur.nl/en/Research-Results/Research-Institutes/plant-research/Biometris-1/Software-Service/Download-MapChart.htm
目前最新版本的Mapchart 2.32,内置6个mct格式案例数据。
下载时需进行注册,提交邮箱信息方便license file文件下载,并将下载的MapChart.lic添加到安装路径,以免影响软件使用。
准备数据
整理染色体的名称、长度信息,以及染色体上需要标注的基因的位置信息,只需要填写基因的起始位置即可,例如:

注意:
1、每一条染色体都是以“chrom”开始表示(以“group”表示linkage group,注意区别),各成分之间以空格分开即可(不拘多少个空格,但是为了数据显示美观,最好保持一致),每一个chrom下的基因都是属于该对应的染色体上的基因;
2、基因之间有先后顺序,严格按照在染色体上的先后顺序排列;
3、各行之间可以有空行,如需注释掉某行内容,行首输入半角下的“;”,此行内容将不会在图片中显示;
4、如需添加第二条染色体数据,可以直接在数据后面添加,格式同上。
基础绘图
软件在正常安装完成后,打开显示的界面如下图:
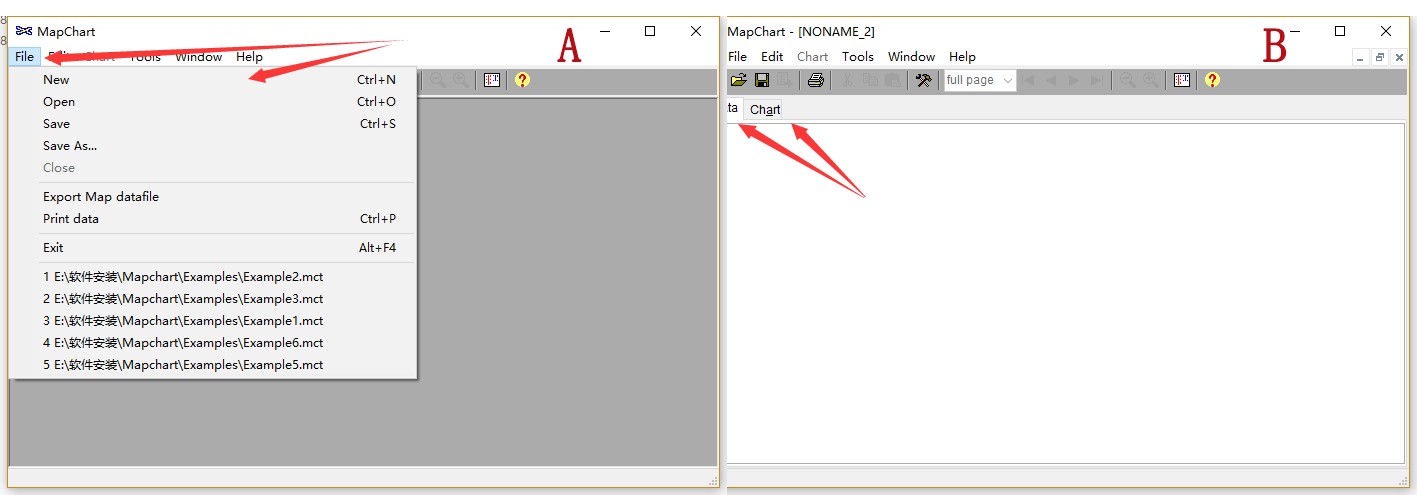
开始绘图,点击File->New(下图A),打开对应的绘图操作界面(下图B),在弹出的操作界面中包括了两个页面,其一是Data,其二为Chart。Data是进行数据填写、调整修改的窗口、而Chart根据Data直接显示图片。
譬如,将前面准备的数据直接复制粘贴到Data中(下图A),或者通过File-->open导入前面准备的文件(建议文本格式txt文件),点击Chart,可以在此页面查看到最原始的图片(下图B)。注意,如绘制染色体较多,Chart页面中的图片可能会有多页。

细节调整
在Data页面中,可以在每行基因信息的后方添加设置,包括字体类型、文字大小、颜色。譬如下图A中在基因A 后添加的信息:
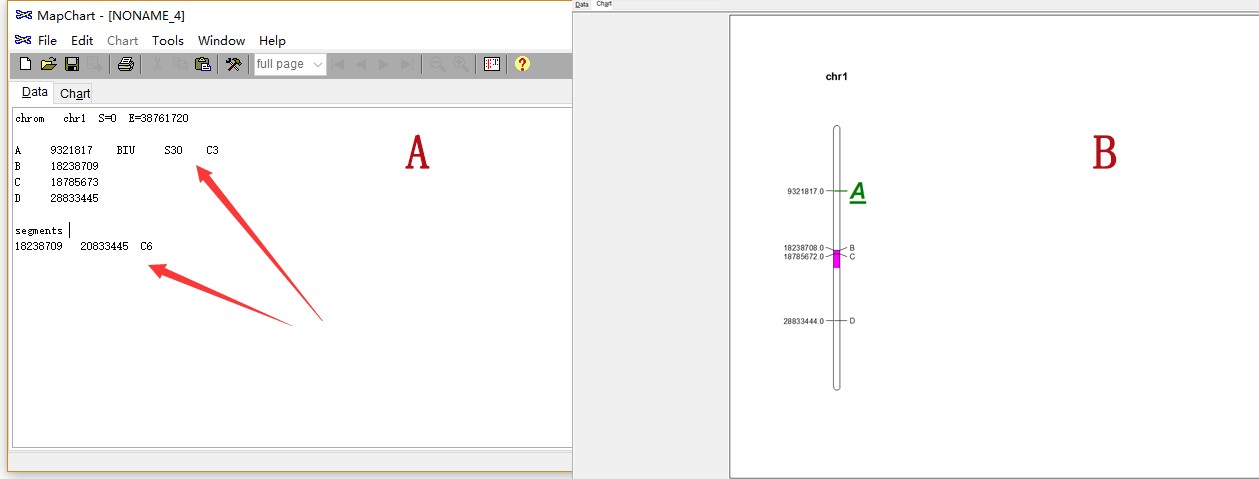
1、 BIU,控制字体类型,B表示加粗,I表示斜体,U表示下划线,这些可以任意组合;
2、 S30,S控制大小,30对应30pt
3、 C3,C对应颜色,C3具体是绿色。
此外,还可以对整条染色体区域颜色进行填充,譬如在该chrom下填写“segments”, (另起一行)起始位置+颜色控制。各成分之间同样是以空格分开(上图A),之后可以在Chart中查看到染色体图片细节的变化(上图B)。
同时,通过Tools->chart options等,设置图片其他的一些属性,包括整个图片背景、图片titile、位置等调整。

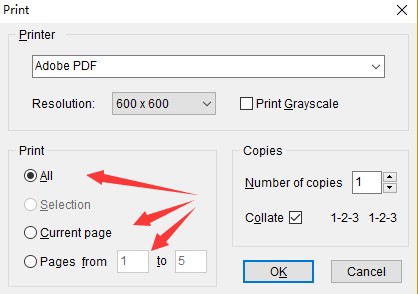
在完成图片的设置之后,可以通过Chart ->print,选中Adobe PDF(需提前安装此虚拟打印机),选择需要打印的page从而将文件保存成pdf。或者直接Char->copy page,进入Ai粘贴即可。
其他
Mapchart的设置涉及的细节处理较多,这些具体设置方法可以通过软件内置的案例数据进行学习。软件内置了六套数据(可在安装路径下Examples文件夹中找到),通过File->Open可以打开这些数据,之后借助每套数据的Data和Chart页面来的理解设置和图片显示具体的对应关系,从而去学习每一个设置的概念。

推荐课程:
基因家族分析实操课程、基因家族文献思路解读
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、perl入门到精通、perl语言高级、R语言入门、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、GEO芯片数据标准化、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读
- 发表于 2018-08-30 18:38
- 阅读 ( 44202 )
- 分类:软件工具
你可能感兴趣的文章
- Mapchart绘制基因染色体位置图时加标尺 2865 浏览
- CRISPR技术将铜绿假单胞菌研究推向新篇章-源井生物 2078 浏览
- Mapchart位置坐标小数点后数字调整 3136 浏览
- Mapchart 添加标尺 5571 浏览
- 收藏贴-基因/蛋白功能注释、富集分析在线工具详解 6173 浏览
